
- DAZ.online
- News
- Pharmazie
- Wie sieht das ...
Max-Planck-Institut und PEI
Wie sieht das Spikeprotein von SARS-CoV-2 aus?
Stuttgart - 19.08.2020, 11:45 Uhr
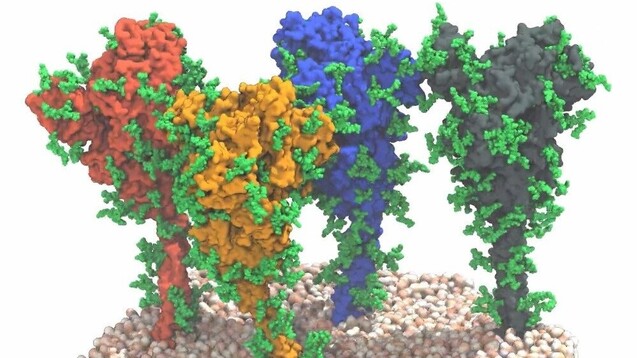
Vier Spikeproteine auf der Membranoberfläche von SARS-CoV-2: Forscher des PEI und MPI haben die Beweglichkeit der Spikeproteine gezeigt. Das kann für die Impfstoffentwicklung von COVID-19-Impfstoffen bedeutsam sein. (Quelle: MPI f. Biophysik / von Bülow, Sikora, Hummer)
Über Spikeproteine (S) auf seiner Oberfläche infiziert SARS-CoV-2 menschliche Zellen. Wissenschaftler des Paul-Ehrlich-Institutes haben nun gemeinsam mit anderen Forschern diese Spikeproteine analysiert – sie scheinen unvermutet beweglich zu sein. Was bedeutet das?
Das neuartige Coronavirus SARS-CoV-2 benötigt Spikeproteine (S) auf seiner Oberfläche, um an spezifische Rezeptoren auf der Oberfläche menschlicher Zellen zu binden und über diesen Weg die Zellen zu infizieren. Neben auf RNA basierenden Impfstoffen stehen diese Spikes, die stachelförmigen Strukturen, im Zentrum des Interesses bei der Entwicklung von COVID-19-Impfstoffen. Als Impf-Antigen sollen sie eine schützende Immunantwort provozieren. Wie genau diese Spikeproteine aussehen, ihre strukturelle Analyse, haben sich Wissenschaftler um Prof. Jacomine Krijnse Locker, Leiterin der Loewe-Forschungsgruppe „Elektronenmikroskopie von Pathogenen“, und Priv.-Doz. Michael Mühlebach des Paul-Ehrlich-Instituts (PEI) intensiv angeschaut – im Verbund mit weiteren Forschenden des Europäischen Laboratoriums für Molekularbiologie (EMBL, European Molecular Biology Laboratory), des Max-Planck-Instituts für Biophysik sowie dem Institut für Biophysik der Goethe-Universität Frankfurt/Main.
Wichtig für die COVID-19-Impfstoff-Forschung
Ziel war es, „Erkenntnisse sowohl für die Impfstoffe als auch für die Entwicklung wirksamer Therapeutika zur Behandlung infizierter Patientinnen und Patienten zu gewinnen“, teilt das PEI mit. Gerade die räumlichen Strukturen – die für Prozesse wie beispielsweise das Eindringen in die Zielzellen wichtig sind – seien hierbei von großer Bedeutung. Ihre Ergebnisse sind seit dem 18. August online bei „Science“ veröffentlicht.
Beweglichkeit der vier Spikeproteine auf der Membranoberfläche von SARS-CoV-2. (Quelle: Sören von Bülow, Mateusz Sikora, Gerhard Hummer, MPI für Biophysik)
„Die Kombination modernster Technologien der Kryo-Elektronentomografie, Subtomogramm-Mittelung und Molekulardynamik-Simulationen ermöglichen die strukturelle Analyse von Molekülstrukturen in natürlicher Umgebung und mit nahezu atomarer Auflösung“, so das PEI. Der Mitteilung zufolge nutzten die Wissenschaftler SARS-CoV-2-Partikel, die aus dem Überstand infizierter Zellen gewonnen wurden. Mithilfe modernster Elektronenmikroskopie am EMBL wurden 266 Kryo-Tomogramme von etwa 1.000 verschiedenen Viren erstellt, die jeweils durchschnittlich 40 Spikes auf ihrer Oberfläche trugen. So konnten von insgesamt 40.000 Spikes wichtige Strukturinformationen gewonnen werden.




















0 Kommentare
Das Kommentieren ist aktuell nicht möglich.