
- DAZ.online
- DAZ / AZ
- DAZ 44/2017
- Lesezeichen im Buch des ...
Epigenetik
Lesezeichen im Buch des Lebens
Gezielte Modifikationen machen die Genexpression der Krebszellen angreifbar
Epigenetische Informationen stellen eine Art Gedächtnis der Zelle dar, sie bilden jedoch – im Gegensatz zum Genom – kein starres System, sodass eine Anpassung an verschiedene Entwicklungsstadien und die Reaktion auf äußere Einflüsse möglich ist. Ein eindrucksvolles Beispiel hierfür ist die Entwicklung von Bienenlarven in einem Bienenstock, die alle die gleiche DNA-Sequenz aufweisen und genetisch wie Klone sind. Abhängig von der Ernährung entwickeln sich aus ihnen völlig unterschiedliche Phänotypen: Wird eine Larve mit Pollen gefüttert, entwickelt sie sich zu einer Arbeiterbiene, erhält sie Gelée royale, entsteht eine Bienenkönigin. Diese massiven Unterschiede können auf epigenetische Mechanismen zurückgeführt werden, welche durch die Ernährung reguliert werden [2].
Aus der Beeinflussbarkeit epigenetischer Faktoren ergeben sich therapeutische Möglichkeiten bei zahlreichen Erkrankungen, welche im Zusammenhang mit epigenetischer Fehlregulation stehen. Beispielsweise kann die verminderte Expression eines Tumorsuppressorgens zur Krebsentstehung beitragen. Weitere Erkrankungen mit epigenetischer Beteiligung sind verschiedene kardiovaskuläre, metabolische sowie neurologische Erkrankungen [3]. Im Gegensatz zu genetischen Mutationen sind solche epigenetischen Anomalien (sogenannte Epimutationen) reversibel, sodass die Fehler prinzipiell durch die Gabe von Arzneistoffen wieder korrigiert werden können.
Eigene Forschung
Unsere Forschungsschwerpunkte sind die Synthese von Hemmstoffen Histon-modifizierender Enzyme, die Entwicklung von Assaymethoden zur Bestimmung der Hemmpotenz dieser Inhibitoren sowie die Validierung der In-vitro-Ergebnisse auf zellulärer Ebene. Wir bearbeiten dabei Zink-abhängige Histon-Desacetylasen (HDACs), NAD+-abhängige HDACs (Sirtuine), Acetyl- bzw. Methyltransferasen, verschiedene Lysin-Demethylasen wie LSD1 und Lese-Proteine („readers“) wie Spindlin1.
Ein Teil der biologischen Arbeiten wird mit verschiedenen Kooperationspartnern durchgeführt. Besonders wichtig sind dabei der Sonderforschungsbereich „Medizinische Epigenetik“ (www.sfb992.uni-freiburg.de) und ein EU-gefördertes Konsortium zur Entwicklung epigenetischer Hemmstoffe gegen parasitäre Tropenerkrankungen wie Bilharziose oder Malaria.
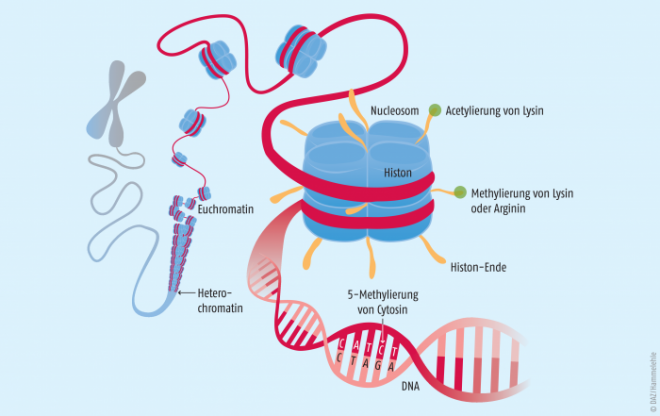
Nucleosom und Histone
Der epigenetischen Regulation liegen verschiedene molekulare Mechanismen zugrunde: RNA-Interferenz, Modifikation einzelner DNA-Basen sowie Histon-Modifikationen. Histone sind basische Strukturproteine, die dazu dienen, den 2 m langen DNA-Strang so zu kompaktieren, dass er im 10 bis 100 µm großen Zellkern Platz findet, aber dennoch für das Ablesen im Rahmen der Transkription zugänglich bleibt. Dazu bilden jeweils acht Histone (jeweils zwei Kopien der Histone H2A, H2B, H3 und H4) einen Komplex, um den ein etwa 147 Basenpaare langer DNA-Abschnitt gewunden ist [4]. Diese Einheit wird als Nucleosom bezeichnet. Die Nucleosomen werden durch Linker-Histone weiter verdichtet, sodass der Chromatinfaden entsteht, der in seiner vollständig kondensierten Struktur die Chromosomen ausbildet (Abb. 1).
Die Histone üben nicht nur eine Gerüstfunktion aus, sondern tragen wesentlich zur Transkriptionsregulation bei: Sie bestimmen mit, welche Gene abgelesen bzw. stillgelegt werden. Die Regulation erfolgt über das Anfügen kleiner Molekülgruppen vor allem an den N-terminalen Enden der Histone. Die beiden am besten untersuchten Modifikationen sind
- die Acetylierung von Lysinresten (an der freien Aminogruppe) und
- die Methylierung von Lysin- oder Argininresten (Abb. 1).
Des Weiteren sind Phosphorylierung, Ubiquitinylierung und ADP-Ribosylierung bekannt [5].
Histon-Code
Die Histon-Modifikationen werden in Analogie zum genetischen Code auch als Histon-Code bezeichnet [6]. Jedoch ist es nicht möglich, einzelnen Modifikationen genaue Funktionen zuzuordnen, da die Effekte einzelner Markierungen auch davon abhängen, welche Modifikationen in der Umgebung vorliegen. Das Zusammenspiel der Histon-Modifikationen ähnelt also eher einer komplexen Sprache, in der die Bedeutung einzelner Wörter vom Kontext abhängig ist, als einem eindeutigen Code, wie er auf der DNA vorliegt.
DNA-Methylierung und -Demethylierung
Die DNA-Methylierung (und RNA-Methylierung) erfolgt am Kohlenstoffatom C-5 von Cytosin, und hier bevorzugt an Cytosin-Guanin-Dinucleotiden (CpGs), die gehäuft in Promotorregionen vorkommen (Abb. 1). Die Methylgruppen beeinflussen die Basenpaarung von Cytosin mit Guanin nicht, sodass der genetische Code unverändert bleibt, sie können jedoch Wechselwirkungen mit sogenannten Lese-Proteinen („readers“) bewirken, die erkennen, ob eine Methylierung am C-5 vorliegt. 5-Methyl-CpG-bindende Proteine in der Promotorregion bewirken eine Stilllegung des zugehörigen Gens. Dieser Mechanismus kommt z. B. bei der Zelldifferenzierung zum Tragen, indem die für den entstehenden Zelltyp nicht relevanten Gene stillgelegt werden [7].
Die Methylierung erfolgt durch DNA-Methyltransferasen, die zur Übertragung der Methylgruppe den Cofaktor S-Adenosylmethionin nutzen. Es werden die Subtypen DNMT1, -2, -3a und -3b unterschieden [8].
Die Demethylierung erfolgt auf indirektem Weg: Nach der Oxidation zu Hydroxymethyl-, Formyl- und schließlich Carboxyl-Cytosin; durch TET-Dioxygenasen kann die modifizierte Base durch DNA-Reparaturenzyme erkannt und durch Cytosin ersetzt werden [9]. Aber auch Mechanismen einer direkten Demethylierung nach der Oxidation durch TET-Enzyme werden diskutiert.
Eine Fehlregulation der DNA-Methylierung kann die Krebsentstehung begünstigen, z. B. indem Tumorsuppressorgene durch eine Hypermethylierung der Promotorregion stillgelegt werden. Eine Reaktivierung dieser Gene durch die Gabe von DNMT-Hemmstoffen stellt eine mögliche therapeutische Intervention dar (s. u.).
Histon-Acetylierung und -Desacetylierung
Die Histon-Acetylierung erfolgt an der freien Aminogruppe der Aminosäure Lysin, vor allem im Bereich der N-terminalen Histon-Enden. Die Acetylgruppe vermindert die Wechselwirkung der Histone mit dem negativ geladenen DNA-Rückgrat. Dies führt zu einer Abnahme der Packungsdichte und damit zu einer Auflockerung der Chromatinstruktur. Das als Euchromatin bezeichnete aktive Chromatin ist leicht für Transkriptionsfaktoren zugänglich, sodass Gene verstärkt abgelesen werden können. Desacetylierte Histone führen zum kompakten Heterochromatin, dessen Transkriptions-Aktivität gering ist. Des Weiteren beeinflusst der Acetylierungsgrad die Wechselwirkung der Histone mit Lese-Proteinen („readers“), welche ihrerseits die Transkription regulieren können.
Histon-Acetyltransferasen (HATs), die Acetyl-CoA als Cofaktor nutzen, katalysieren die Acetylierung, während Histon-Desacetylasen (HDACs) die Desacetylierung bewirken [10]. Die 18 humanen HDAC-Subtypen werden anhand ihrer Struktur vier Klassen zugeordnet:
- HDACs der Klassen I, II und IV enthalten ein Zink-Ion, welches die Amid-Hydrolyse katalysiert. Ihr Wirkmechanismus ist analog zu dem ebenfalls Zink-abhängigen Angiotensin Converting Enzym (ACE). Wie ACE-Hemmstoffe (Captopril u. a.), die an das Zink-Ion binden, besitzen HDAC-Inhibitoren Zink-bindende Strukturen.
- HDACs der Klasse III werden als Sirtuine bezeichnet; in ihrer Struktur und ihrem Wirkmechanismus unterscheiden sie sich deutlich von den übrigen drei Klassen. Die Katalyse erfolgt mithilfe des Cofaktors NAD+. Neben der Lysin-Desacetylierung können Sirtuine auch die Abspaltung anderer Acylreste wie Butyrat oder Succinat katalysieren, was weitere Regulationen ermöglicht [11].
Sowohl HATs als auch HDACs können auch Nicht-Histon-Proteine modifizieren (z. B. Tubulin als Bestandteil der Mikrotubuli oder das Tumorsuppressorprotein p53), weshalb teilweise die allgemeineren Bezeichnungen Lysin-Acetyltransferasen und Lysin-Desacetylasen (KATs und KDACs) in der Literatur zu finden sind (K = Symbol für Lysin).
Störungen im Gleichgewicht zwischen Acetylierung und Desacetylierung spielen bei Krebserkrankungen eine Rolle. Eine erhöhte HDAC-Aktivität vermindert die Expression verschiedener Tumorsuppressorgene, was zur Krebsentstehung beitragen kann. HDAC-Inhibitoren können der verminderten Expression entgegenwirken [12].
Histon-Methylierung und -Demethylierung
Die Histon-Methylierung erfolgt an Lysin- und Argininresten. Arginin kann ein bis zwei Methylgruppen tragen, Lysin bis zu drei. Daraus ergibt sich eine Vielzahl von Kombinationen, welche eine komplexe Regulation ermöglichen. Die Modulation der Transkriptionsrate erfolgt bei der Histon-Methylierung überwiegend durch Wechselwirkungen mit Lese-Proteinen; dabei wird die Packungsdichte des Chromatins kaum beeinflusst. Die Lese-Proteine erkennen oft spezifische Modifikationsmuster, sodass Effekte sowohl von der Position der methylierten Aminosäure und der Anzahl der Methylreste als auch von der Lage weiterer Histon-Modifikationen beeinflusst werden. So kann eine Methylierung sowohl die Aktivierung als auch die Repression einer Transkription vermitteln. Histon-Methyltransferasen (HMTs) katalysieren die Methylierung, Histon-Demethylasen (HDMs) die Demethylierung.
Wie die (Des-)Acetylierung ist auch die (De-)Methylierung nicht auf Histone beschränkt, sondern kann auch an anderen Proteinen wie p53 erfolgen; zudem werden auch epigenetische Enzyme methyliert oder acetyliert. Deshalb werden immer häufiger die allgemeineren Bezeichnungen KMT und KDM für die Lysin-modifizierenden Enzyme (vgl. KATs und KDACs) bzw. PRMT für die Protein-Arginin-Methyltransferasen verwendet (R = Symbol für Arginin); Arginin-Demethylasen wurden in vivo bislang noch nicht eindeutig identifiziert.
Die Histon-Demethylasen werden in zwei Gruppen unterteilt,
- die Flavin-Adenin-Dinucleotid(FAD)-abhängigen Lysin-spezifischen Demethylasen LSD1 und -2 sowie
- die Eisen- und α-Ketoglutarat-abhängigen Demethylasen KDM2 bis -7.
Der katalytische Mechanismus von LSD1 und -2 entspricht dem der Monoaminoxidasen MAO A und B, welche eine Sequenzhomologie von etwa 20% zu LSD aufweisen, sodass einige MAO-Hemmstoffe auch als LSD-Hemmstoffe wirken (s. u.). Basierend auf der Struktur zugelassener MAO-Hemmstoffe konnten selektive LSD1-Inhibitoren entwickelt werden [13].
Epigenetik in der Krebstherapie
DNMT-Inhibitoren
Die beiden ersten zur Therapie zugelassenen epigenetischen Medikamente sind die Nucleosidanaloga Azacitidin (5-Azacytidin, Vidaza®) und Decitabin (Dacogen®; Tab. 1, Abb. 2). Nach dem Einbau in die DNA führen sie zur irreversiblen Hemmung von DNA-Methyltransferasen und können damit der Hypermethylierung von Tumorsuppressorgenen entgegenwirken. Neben dem Eingriff in epigenetische Regulationsmechanismen wurden auch allgemein zytotoxische Wirkungen für die beiden Substanzen beschrieben. So wird Azacitidin auch in RNA inkorporiert und stört die RNA-Prozessierung.
Zielstruktur |
Hemmstoff |
Indikation* |
|---|---|---|
DNA-Methyltransferasen (DNMTs) |
Azacitidin (Vidaza®)
Decitabin (Dacogen®)
|
MDS, CMML, AML
AML
|
Histon-Desacetylasen (HDACs) |
Vorinostat (Zolinza®)
Belinostat (Beleodaq®)
Panobinostat (Farydak®)
Valproinsäure
|
kutanes T-Zell-Lymphom
peripheres T-Zell-Lymphom
multiples Myelom
MDS (in klinischer Prüfung)
|
Histon-Demethylasen (HDMs) |
Tranylcypromin |
AML (in klinischer Prüfung) |
* AML = akute myeloische Leukämie, CMML = chronische myelomonozytäre Leukämie, MDS = myelodysplastisches Syndrom | ||
Azacitidin ist zur Behandlung maligner hämatologischer Erkrankungen wie myelodysplastischem Syndrom, chronischer myelomonozytärer Leukämie und akuter myeloischer Leukämie (AML) zugelassen, wenn eine Stammzelltransplantation nicht möglich ist. Decitabin wird bei AML eingesetzt.
Weitere DNMT-Inhibitoren werden in klinischen Studien untersucht, beispielsweise das Dinucleotid Guadecitabin, welches sich in Phase III der klinischen Entwicklung befindet. Es soll eine längere Wirkdauer als Decitabin besitzen und wird neben der Therapie hämatologischer Erkrankungen auch auf seine Wirksamkeit in soliden Tumoren untersucht.
HDAC-Inhibitoren
Eine zweite Gruppe epigenetischer Medikamente in der Krebstherapie sind HDAC-Inhibitoren. Die FDA hat 2006 mit Vorinostat (Zolinza®; Tab. 1, Abb. 2) den ersten Hemmstoff Zink-abhängiger HDACs zugelassen; von der EMA erhielt er den Status eines Orphan Drug zur Behandlung des kutanen T‑Zell-Lymphoms. Wichtiger struktureller Bestandteil ist eine Hydroxamsäuregruppe, welche die Bindung an das Zink-Ion in der HDAC vermittelt.
Vorinostat ist zur Behandlung zugelassen, wenn bereits zwei systemische Therapien erfolglos waren oder ein Rückfall vorliegt. Große Aufmerksamkeit erhielt der Wirkstoff durch seinen möglichen Einsatz in der HIV-Therapie. Die Behandlung HIV-infizierter T‑Zellen mit Vorinostat reaktiviert latente Viren, die von konventionellen HIV-Therapeutika nicht erfasst werden. Durch die Reaktivierung dieser Viren soll eine vollständige Eradikation befallener Zellen und damit die Heilung erreicht werden. Bislang konnte in klinischen Studien die Reaktivierung der latenten Viren gezeigt werden, jedoch führte sie nicht zur erhofften Reduktion der Anzahl infizierter Zellen. Um dieses Ziel zu erreichen, sollen geeignete Kombinationspartner für die Therapie mit HDAC-Inhibitoren identifiziert werden [14].
Mit Belinostat (Beleodaq®) und Panobinostat (Farydak®; Tab. 1, Abb. 2) hat die FDA zwei weitere Pan-HDAC-Inhibitoren mit Hydroxamsäurestruktur zugelassen. Belinostat erhielt 2012 von der EMA den Status als Orphan Drug zur Behandlung des peripheren T‑Zell-Lymphoms, Panobinostat erlangte 2015 die Zulassung zur Therapie des multiplen Myeloms in Kombination mit dem Proteasom-Inhibitor Bortezomib und Dexamethason.
Das zyklische Peptid Romidepsin (Istodax®) ist ein Klasse-I-selektiver HDAC-Inhibitor, der 2009 von der FDA zur Behandlung des kutanen und peripheren T‑Zell-Lymphoms zugelassen wurde. Die EMA verweigerte die Zulassung aufgrund ungenügenden Nachweises des klinischen Nutzens.
Häufige Nebenwirkungen, die bei der Behandlung mit HDAC-Inhibitoren auftreten, sind Durchfall, Erbrechen, Fatigue und Thrombozytopenie. Die hohe Nebenwirkungsrate, die mit steigender Dosierung zunimmt, steht einer breiten Anwendung der HDAC-Inhibitoren bei verschiedenen Krebserkrankungen im Wege. Deshalb werden
- einerseits Kombinationstherapien mit anderen Krebsmedikamenten getestet, um die Dosierung der einzelnen Wirkstoffe möglichst zu verringern und die Therapieeffizienz zu erhöhen,
- andererseits zahlreiche neue HDAC-Inhibitoren entwickelt, die eine Selektivität für einzelne HDAC-Klassen oder -Subtypen aufweisen, wovon weniger Nebenwirkungen erhofft werden.
Am weitesten fortgeschritten in der klinischen Prüfung sind die selektiven HDAC-Inhibitoren
- Entinostat zur Behandlung von fortgeschrittenem Brustkrebs,
- Pracinostat zur Therapie der akuten myeloischen Leukämie und
- Givinostat zur Therapie des multiplen Myeloms und der juvenilen idiopathischen Arthritis.
In China wurde Tucidinostat (Chidamide, Epidaza®), das selektiv die HDAC-Subtypen 1 bis 3 hemmt, zur Behandlung des T‑Zell-Lymphoms zugelassen. Es steht noch aus, ob daraus ein klinischer Vorteil resultiert [15].
Mit Ricolinostat wird erstmals ein selektiver Hemmstoff des Subtyps HDAC6 in klinischen Studien getestet. In der Kombination mit dem Proteasomhemmer Bortezomib und Dexamethason wurde ein Ansprechen von 37% der Patienten mit Multiplem Myelom beobachtet [16].
Auch Inhibitoren von Lysin-Methyltransferasen (KMTs) befinden sich in klinischen Studien zur Behandlung von Krebserkrankungen [19]. Beispiele sind der DOT1L-Hemmstoff Pinomestostat (bei MLL-r) und der EZH2-Inhibitor Tazemestostat (u. a. bei Non-Hodgkin-Lymphomen).
Neue Indikationen für alte Arzneimittel
Eine weitere Strategie in der Entwicklung neuer Therapieoptionen stellt das „drug repositioning“ dar, d. h. die Anwendung bereits zugelassener Medikamente in neuen Indikationen. Vorteile dieses Ansatzes sind, dass mögliche Nebenwirkungen bereits bekannt sind und eine massive Einsparung von Zeit und Ressourcen im Vergleich zur Neuentwicklung eines Medikaments möglich ist. Beispiele im Bereich der Epigenetik sind das Antiepileptikum Valproinsäure, das in hoher Dosierung HDACs hemmt, und das Antihypertensivum Hydralazin, welches ein DNMT-Hemmstoff ist. Die kombinierte Anwendung beider Medikamente zeigte in klinischen Studien vielversprechende Wirkungen bei der Therapie des myelodysplastischen Syndroms [17].
Ein weiteres Beispiel für drug repositioning stellt das Antidepressivum Tranylcypromin dar. Dieses als MAO-Inhibitor entwickelte Medikament zeigt eine inhibitorische Wirkung auf Lysin-spezifische Histon-Demethylasen wie LSD1. Es wird in klinischen Studien als Therapeutikum bei akuter myeloischer Leukämie getestet, da LSD1 in Leukämiezellen überexprimiert wird. Diese Überexpression ist für die Resistenz der Zellen gegenüber all-trans-Retinsäure (ATRA) verantwortlich, die eine Differenzierung der Leukämiezellen bewirken kann. Durch Behandlung mit Tranylcypromin kann diese Resistenz aufgehoben werden, sodass die Zellen auf eine Behandlung mit ATRA ansprechen [18].
Derivate von Tranylcypromin, welche eine höhere inhibitorische Potenz und Selektivität gegenüber MAO besitzen, befinden sich in Phase I/II der klinischen Entwicklung zur Therapie der AML und des nichtkleinzelligen Bronchialkarzinoms.
Fazit
Die Epigenetik stellt ein faszinierendes Forschungsgebiet mit hoher Relevanz für die Therapie von Krebserkrankungen dar. Medikamente mit epigenetischem Wirkmechanismus stehen seit einigen Jahren zur Therapie zur Verfügung und zahlreiche weitere befinden sich in der klinischen Entwicklung. Durch die Kombination mit anderen Krebsmedikamenten kann die Effektivität der Medikamente noch deutlich gesteigert werden, sodass die Bedeutung epigenetischer Therapieansätze weiter zunehmen wird. Zudem spielen epigenetische Prozesse auch bei vielen Erkrankungen eine wichtige Rolle, zu nennen sind dabei u. a. virale Infektionen, Neurodegeneration, Diabetes und Entzündung. Auch hier sind in den nächsten Jahren spannende neue Ergebnisse zu erwarten, die hoffentlich auch zu neuen therapeutischen Optionen führen werden. |
Literatur
[1] Wagner JM, Jung M. Pharm Unserer Zeit 2010:39(3):176-182
[2] Kucharski R et al. Science 2008;319(5871):1827-1830
[3] Heerboth S et al. Genet Epigenet 2014;6:9-19
[4] Luger K et al. Nature 1997;389(6648):251-260
[5] Kouzarides T. Cell 2007;128(4):693-705
[6] Jenuwein T, Allis CD. Science 2001;293(5532):1074-1080
[7] Meissner A et al. Nature 2008;454(7205):766-770
[8] Okano M, Xie S, Li E. Nature Genet 1998;19(3):219-220
[9] Wu H, Zhang Y. Cell 2014;156(1):45-68
[10] Marmorstein R. Nature Rev Mol Cell Biol 2001;2(6):422-432
[11] Bheda P et al. Annu Rev Biochem 2016;85(1):405-429
[12] Marks PA, Richon VM, Rifkind RA. J Natl Cancer Inst 2000;92(15):1210-1216
[13] Ueda R et al. J Am Chem Soc 2009;131(48):17536-17537
[14] Wu G et al. JCI Insight 2017;2(16)
[15] Xu V, Zhang P, Liu Y. Drugs Today (Barc) 2017;53(3):167-176
[16] Vogl DT et al. Clin Cancer Res 2017;23(13):3307-3315
[17] Candelaria M et al. Encouraging results with the compassionate use of hydralazine/valproate (TRANSKRIP™) as epigenetic treatment for myelodysplastic syndrome (MDS). Ann Hematol; Epub 23.8.2017
[18] Study of Sensitization of Non-M3 AML Blasts to ATRA by Epigenetic Treatment With Tranylcypromine (TCP); https://ClinicalTrials.gov/show/NCT02717884
[19] Morera L, Lübbert M, Jung M. Targeting histone methyltransferases and demethylases in clinical trials for cancer therapy. Clin Epigenet 2016;8:57



























0 Kommentare
Das Kommentieren ist aktuell nicht möglich.